
npj Digital Medicine | 多模态融合学习助力癌症精准预后
癌症患者的预后情况,是制定个性化治疗方案的核心依据,但肿瘤具有高度异质性,给精准评估预后带来了巨大挑战。目前,多模态研究多集中在病理影像与基因组数据,而能反映患者整体健康状况的临床信息,因存在离散、稀疏、维度低等特性,尚未被充分挖掘利用,这也导致传统方法难以精准评估预后风险。
近日,中国科学院深圳先进技术研究院医学成像科学与技术系统全国重点实验室秦文健研究员,提出一种临床信息提示整合的多模态预后预测新框架,通过设计临床文本模板和基础大模型,将结构化临床数据转化为高维语义特征,并通过交叉注意力机制实现病理图像、基因组与临床信息的高效融合,提升癌症生存预测精度。研究成果以"Multimodal deep learning for cancer prognosis prediction with clinical information prompts integration"为题,发表于国际数字医学权威期刊npj Digital Medicine(图1)。
临床信息“沉睡”难题如何破解?
传统多模态研究多集中于病理影像与基因组数据,而年龄、肿瘤分期等临床信息因离散、低维的特性常被忽略。受图像-文本对比学习研究的启发,研究团队设计“临床文本模板”,并借助视觉-语言基础模型编码为高维向量,激活临床数据的深层语义价值。具体而言,研究团队为每一项临床特征设计了围绕关键信息的文本描述模板,并利用GPT-4o mini自动生成多个同义句模板,每个临床特征随机选取一个模板生成文本描述,并通过预训练的文本基础模型进行编码。实验结果表明,当模板数量Nt=1时,不同生存时间患者的临床高维特征在区分性和聚类效果上达到最优(图2a)。病理图像和基因组数据分别通过基础大模型UNI和scFoundation进行特征编码,随后与高维临床特征一同输入SurvPGC模型。该模型采用双路径结构,每一路均基于跨模态双向注意力机制,将病理图像分别与临床信息或基因组数据进行融合(图2d),从而充分挖掘不同模态之间的互补信息。
三大癌种验证:AUC显著提升,风险分层更精准
在性能评估方面,研究将SurvPGC与多种单模态及多模态生存预测模型进行了系统比较。总体而言,多模态模型显著优于单模态模型;其中,SurvPGC在TCGA-LIHC、TCGA-BRCA和TCGA-COADREAD数据集上的C-index分别达到0.701±0.054、0.701±0.057和0.676±0.087,均优于现有性能最优模型。基于Kaplan–Meier曲线的风险分层分析同样表明,SurvPGC在区分高风险与低风险患者方面具有更强的预测能力(图3)。
为进一步分析双路径融合嵌入对模型决策的贡献,研究采用Integrated Gradients(IG)方法量化多模态嵌入对预测结果的影响,并对跨模态注意力矩阵Ac→p(临床-病理学)和Ag→p(基因组-病理学)在病理全切片图像上的分布进行了可视化,以研究基因组和临床信息关注的组织区域。图4展示了来自数据集TCGA-LIHC的典型病例,其中红色表示高关注区域,蓝色表示低关注区域。根据生存时间将病例分为高风险和低风险组,生存时间越短,风险越高。本研究分别根据Ac→p和Ag→p的累积注意力值选择了排名前50的图像块区域。在TCGA-LIHC中(图4),临床和基因组数据的注意力主要集中在WSI中的肿瘤细胞区域。此外,基因组数据还更倾向于关注诸如淋巴细胞和坏死等区域,而这些区域很少被Ac→p选择。然而,基因组数据更容易受到噪声的影响,并可能聚焦于无关的伪影,如临床上的人工马克笔标记或扫描阴影。尽管不同数据集中的临床和基因组数据的主要关注区域有所不同,但总体而言它们是互补的。
中国科学院深圳先进技术研究院博士生侯嘉馨为该研究的第一作者,中国科学院深圳先进技术研究院秦文健研究员为论文通讯作者。该研究工作得到了国家自然科学基金、深港肿瘤影像智能计算分析联合实验室、深圳市基础研究专项自然科学基金、深港澳科技计划以及中国科学院青年创新促进会会员的资助。
图1:文章上线截图

图2:本研究提出的工作流
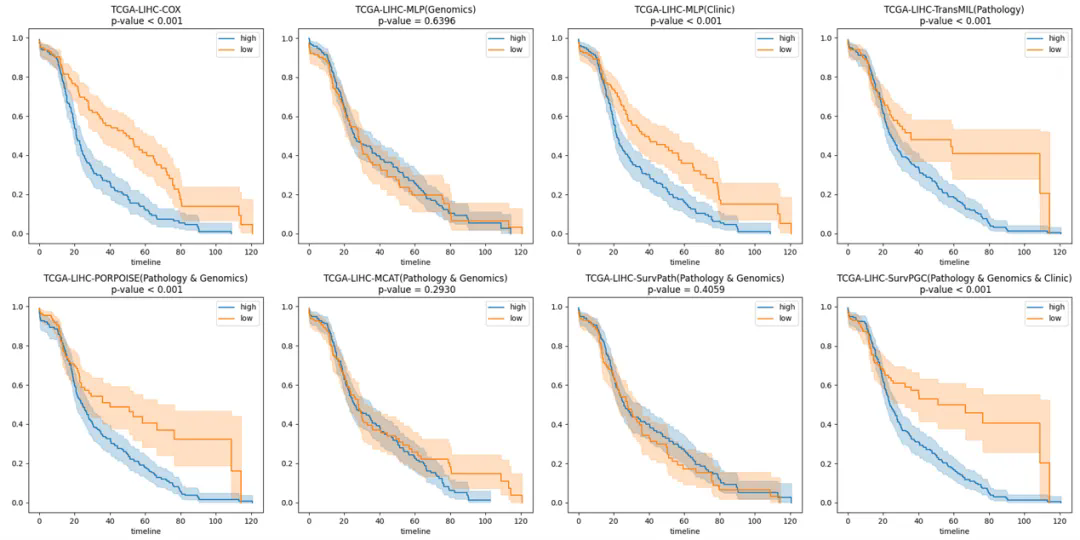
图3:各模型在TCGA-LIHC数据集上的KM曲线
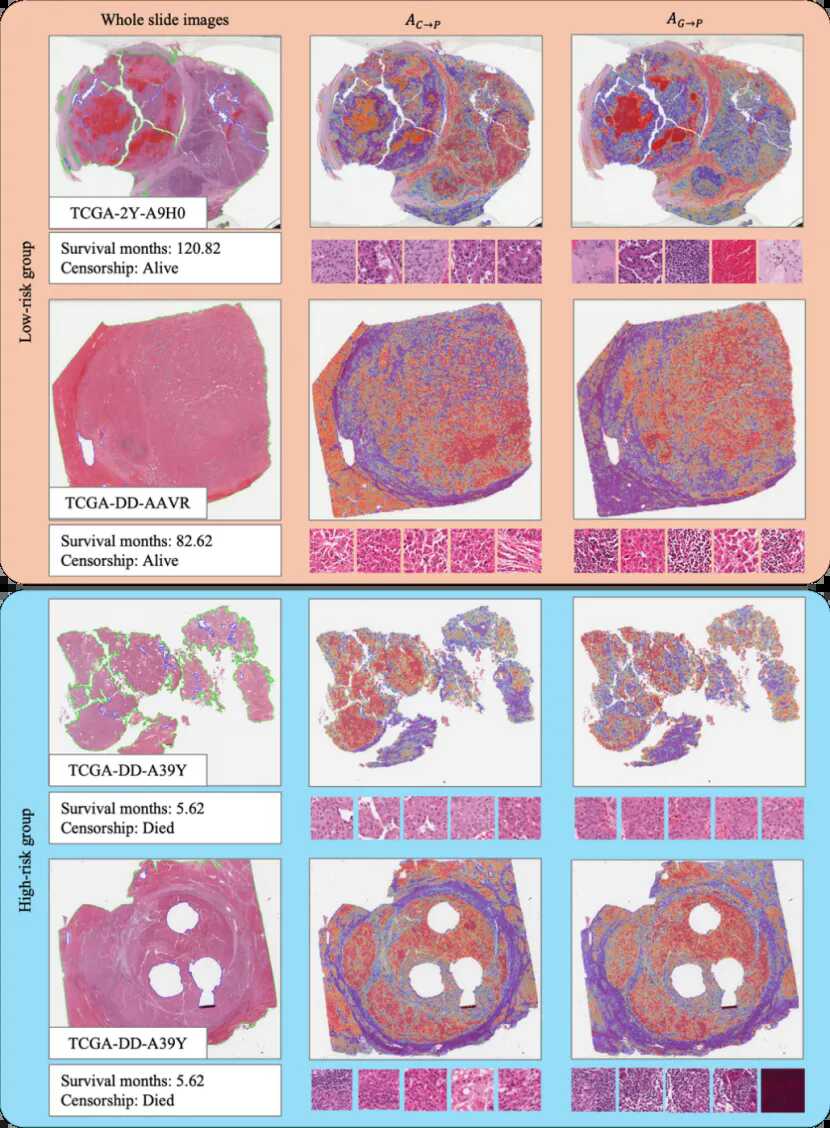
图4:TCGA-LIHC患者的可解释性可视化展示
附件下载: